16 Exercícios
library(ape)
plants.tree <- "((((((((((((((Eudicots), Monocots), Magnoliids), Chloranthales), Autrobaileyales), Nymphaeales), Amborellales),((((Gnetales, Conifers), Gingkoales), Cycadales))),(((Sphenophytes, Marattiales), Filicales),(Ophioglossaceae, Psilophytes))),(Selaginellales, Lycopodiales)),(Bryophyta)),(Anthocerophyta)),(Marchantiophyta)));"
plants.tree <- read.tree(text = plants.tree)
plot(plants.tree)
- Os nomes dos terminais (
tip labels) são numerados em ordem consecutiva começando do 1:
plot(plants.tree)
tiplabels()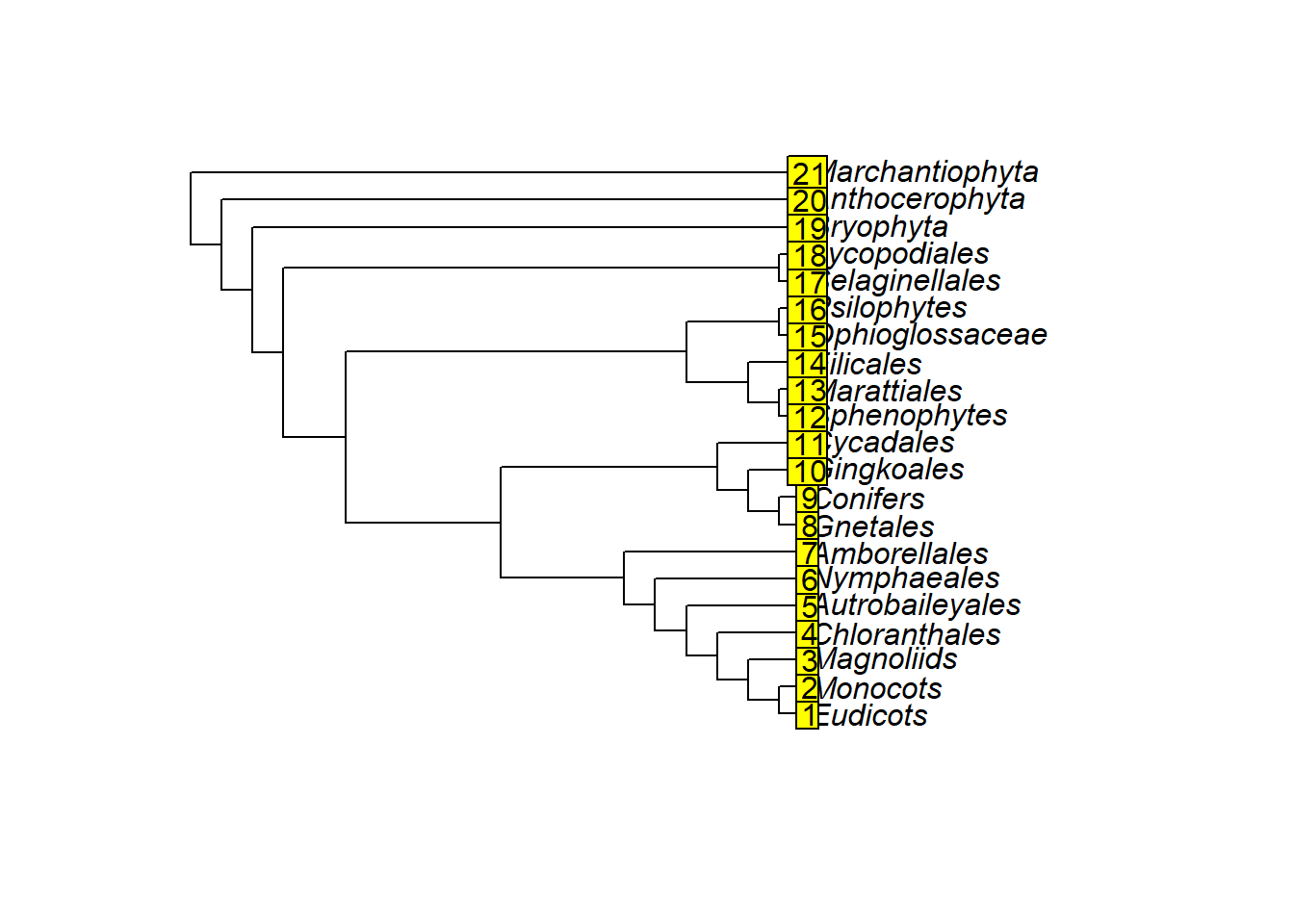
- Os nós (
nodes) são numerados em ordem consecutiva começando do número consecutivo ao número de terminais:
length(plants.tree$tip.label) + 1## [1] 22- Vejam os rótulos (labels) dos nós:
plot(plants.tree)
nodelabels()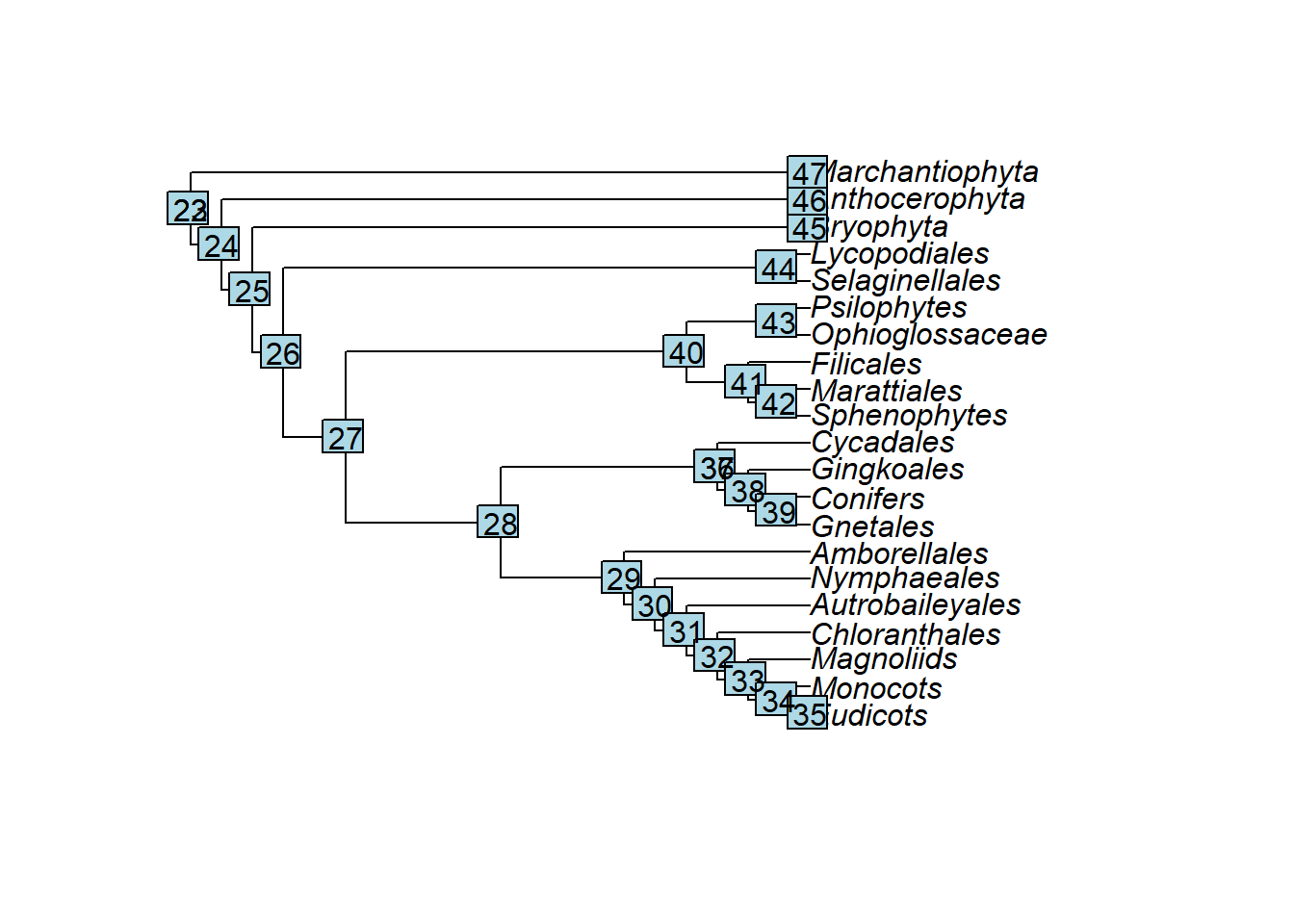
- Vamos extrair os clados com base no número (rótulo) do nó:
tree1 <- extract.clade(phy = plants.tree, node = 29)
plot(tree1)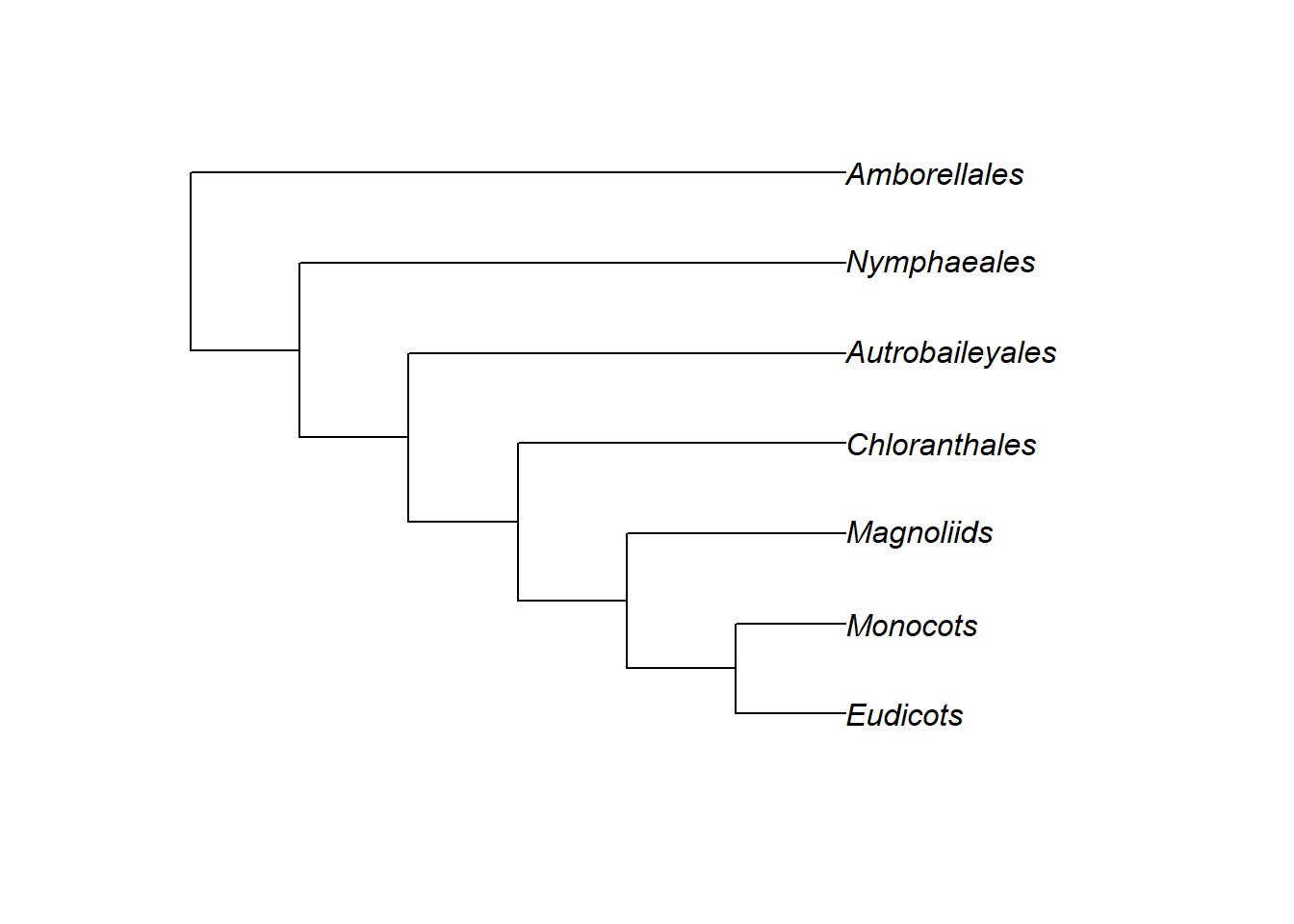
# Tente a seleção do nó interativamente
# tree2 <- extract.clade(plants.tree, interactive = T)
# plot(tree2)Clado das Fixadoras de Nitrogênio (clado CFN) FABA-ROSA-CUCU-FAGA
library(ape)
apg.tree <- read.tree("./files/trees/apg.tree")
nome <- "nitrogenfixing"
busca_clado <- grep(nome, apg.tree$node.label)
apg.tree$node.label[busca_clado]## [1] "nitrogenfixing_to_COM" "nitrogenfixing"if(length(busca_clado) > 1){
node <- max(busca_clado + length(apg.tree$tip.label))
} else {
node <- busca_clado + length(apg.tree$tip.label)
}
tree <- extract.clade(phy = apg.tree, node = node)
plot(tree)
nodelabels(tree$node.label, frame = "none", cex = 0.7)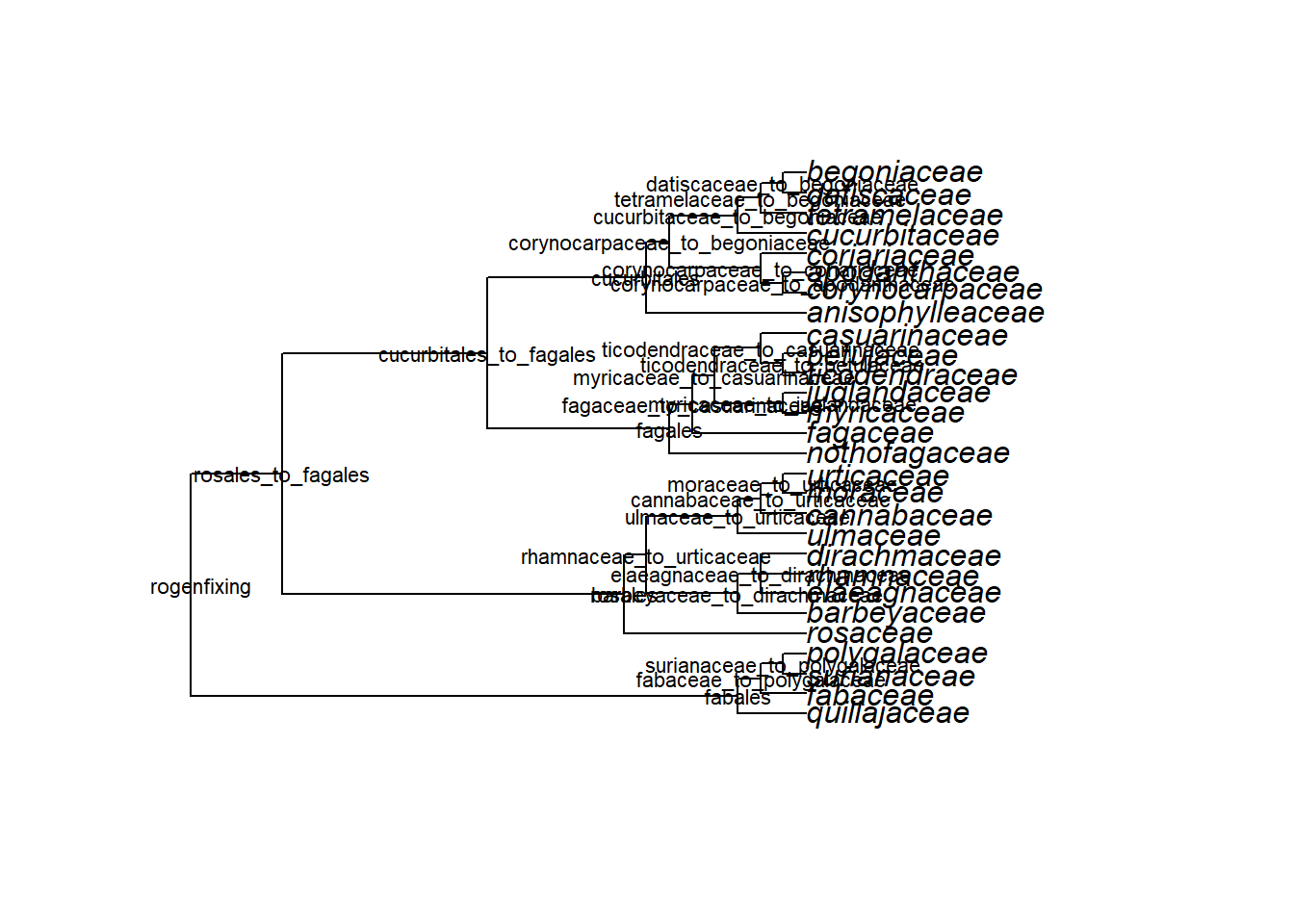
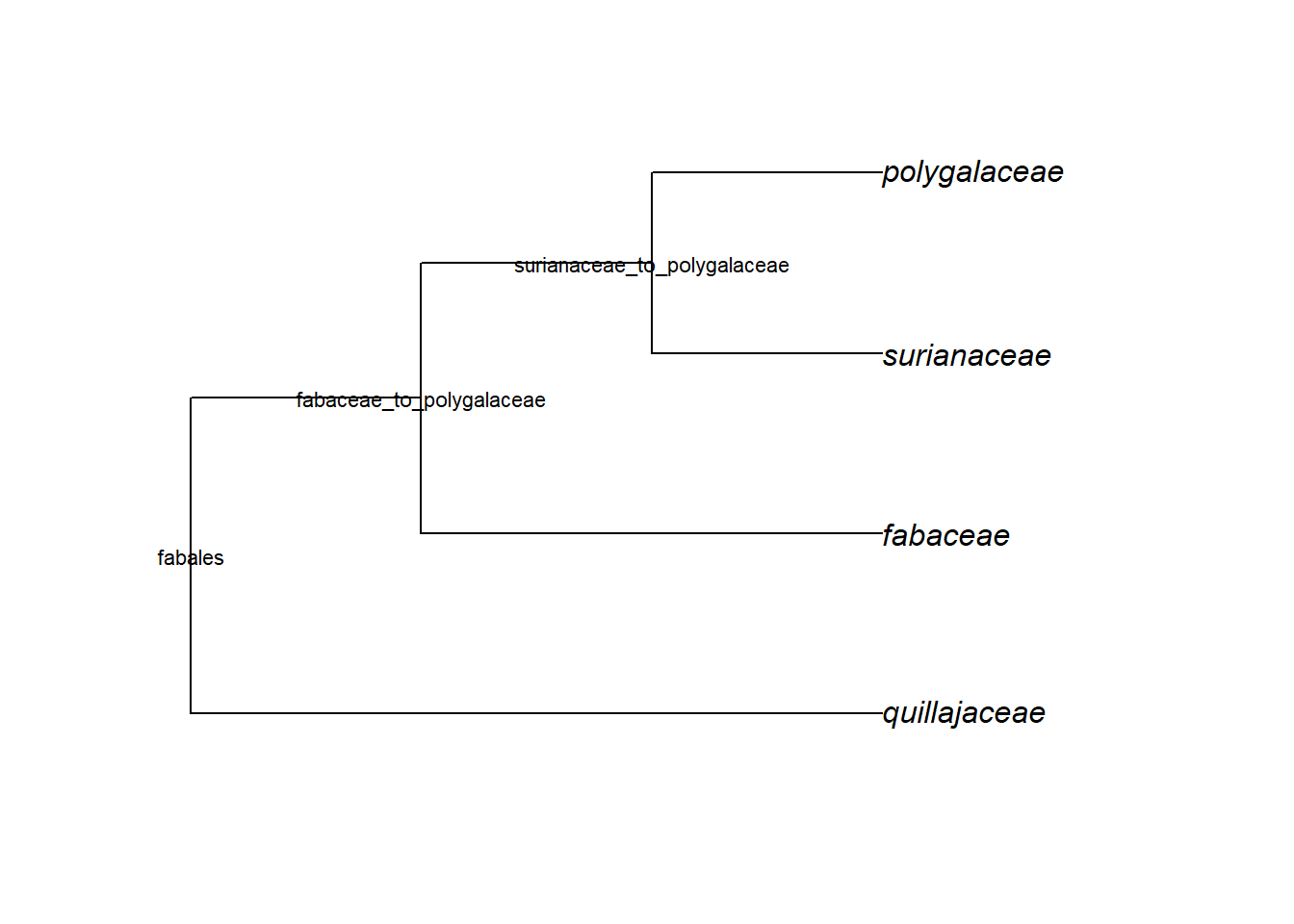
Ordem Fabales
nome <- "fabales"
busca_clado <- grep(nome, apg.tree$node.label)
apg.tree$node.label[busca_clado]## [1] "fabales"if(length(busca_clado) > 1){
node <- max(busca_clado + length(apg.tree$tip.label))
} else {
node <- busca_clado + length(apg.tree$tip.label)
}
tree <- extract.clade(phy = apg.tree, node = node)
plot(tree)
nodelabels(tree$node.label, frame = "none", cex = 0.7)